gnuplotで大量ファイル出力時の高速化
gnuplotで出力区間を変更しながら多数のpngファイル(数百ファイル)に出力していたところ、顕著に遅いことに気付きました。
通常は単一ファイルにプロットすることが殆どだと思われ、このような使われ方はあまり一般的ではないと思うのですが、原因と対策を記します。
遅い原因
そもそもデータ量が膨大なため、時間がかかるのは致し方ないことです。
が、CPUを喰ってません。それでいて、メモリやDISK I/Oも逼迫しておらず、マシンは暇なように見えます。

タスクマネージャで確認すると、最大で25%しかCPUを消費していません。使用マシンは2core/4threadなので、gnuplotはシングルスレッドでしか動作していないようです。
リファレンスを見てもマルチコア/マルチスレッドに関するオプションなどは無さそうです。
(処理内容を察するにマルチスレッドで高速化できる類の処理でもなさそうですし、シングルスレッドでしか動作しないのも特別おかしな話でもないと思います。)
対策
シングルスレッドでしか動作しないなら、gnuplotを複数プロセス立ち上げて並列実行すれば、(オーバーヘッドを無視して)理論上はプロセス数で除した時間にまで所要時間を短縮できるはずです。
対策前は、以下のようなコマンドを出力区間数(約600回)繰り返した単一のスクリプト(plt)ファイルを実行していました。
#出力区間指定
set xrange[FROM:TO]
#出力ファイル指定
set output OUTPUT_N.png
#プロット実行
plot SOURCE.dat with lines
#出力ファイルを外部コマンドで加工
system("EXTERNAL_COMMAND") 前述の通り2core/4thread機のため、スクリプトファイルを4分割して並列実行するのが並列化の上限になりますが、その場合CPU負荷100%で張り付くことが予想され、オーバーヘッドが懸念されます。
そこで、今回はスクリプトファイルを2分割しました。代わりに、以下のように外部コマンド実行部を非同期処理とすることでCPUに余力を持たせることにしました。
#出力ファイルを外部コマンドで加工(非同期)
system("start EXTERNAL_COMMAND") その結果、以下の通り2プロセスのgnuplotが25%ずつ合計50%CPUを消費し、非同期処理で実行される外部コマンドの処理中のみ完全にCPUを食い潰すようにできました。
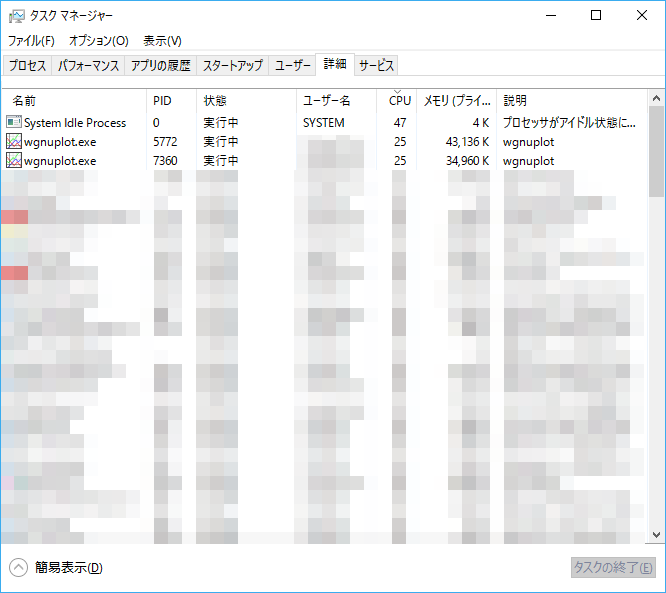
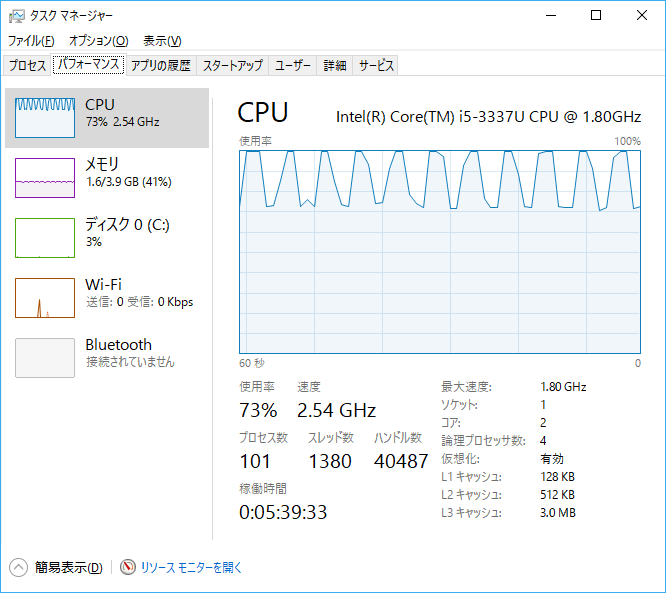
上記対策により、厳密に計測したわけではありませんが、狙い通り所要時間を概ね半分程度に短縮することができました。
なお外部コマンドが実行される都度、起動されたコマンドウインドウにフォーカスが奪われるため、テキストエディタ等の軽い処理であっても、同一のPCで他の作業を行うことは現実的ではありません。
以上。